- Imprimer
- Partager
- Partager sur Facebook
- Partager sur X
- Partager sur LinkedIn
Méthode de modification du génome d'E. coli basée sur CRISPR-Cas12
Cathy Bouchenot, Hans Geiselmann, Noël Scaramozzino, Alexandre Dawid, Stéphan Lacour, Delphine Ropers, Hidde de Jong
Les technologies CRISPR sont devenues des outils de laboratoire standards pour la manipulation génétique dans tous les règnes du vivant. Malgré leur origine bactérienne, le développement des outils CRISPR pour l'ingénierie des bactéries a été plus lent que pour les eucaryotes ; cependant, leur fonctionnement et leur application pour l'ingénierie des génomes ont été démontrés dans une variété de bactéries, et leur adoption s'est généralisée. L'édition génomique par la technologie CRISPR permet la suppression ou l'insertion d'ADN in vivo, soit sur le génome, soit sur des éléments mobiles tels que les plasmides.
Nous mettons en œuvre un système simple basé sur des plasmides pour l'édition du génome (intégration dans le génome de petits et grands fragments d'ADN) chez E. coli en utilisant un système CRISPR-Cas12a et un système de recombinaison λ Red avec une seule étape de clonage (Jervis et al., Microbial Biotechnology, 2021).
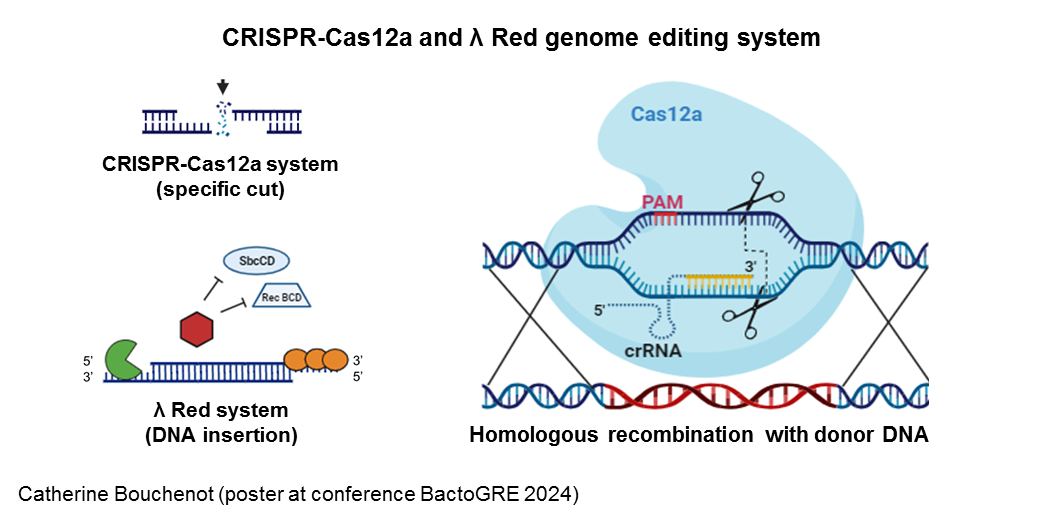
La structure CRISPR est transcrite sous la forme d'un long précurseur (pre-crRNA, non représenté), qui est ensuite transformé par clivage dans les régions répétées pour produire des ARN CRISPR (crRNA) individuels spécifiques à la cible. Ces crRNA dirigent la nucléase Cas12a vers des cibles d'ADN double brin complémentaires (dsDNA), où la nucléase Cas12a crée une cassure double brin (DSB). Les petits ARN guides (crRNA) peuvent diriger le clivage vers toute séquence du génome adjacente à la séquence de 3 pb d'un motif protospacer adjacent (PAM) en modifiant la ou les région(s) espaceur(s). L'import d'ADN donneur (dsDNA) homologue au site de clivage, dans des cellules compatibles avec la recombinaison, permet la réparation de la cassure double brin d'ADN par remplacement de la séquence cible par la séquence du dsDNA au cours du processus. En l'absence de réparation, la cellule meurt, ce qui permet une méthode de sélection « vie–mort ».
- Imprimer
- Partager
- Partager sur Facebook
- Partager sur X
- Partager sur LinkedIn