- Imprimer
- Partager
- Partager sur Facebook
- Partager sur X
- Partager sur LinkedIn
Évolution dirigée par phage display pour sélectionner des ligands ciblés à partir de banques aléatoires de peptides ou d'anticorps
Noël Scaramozzino
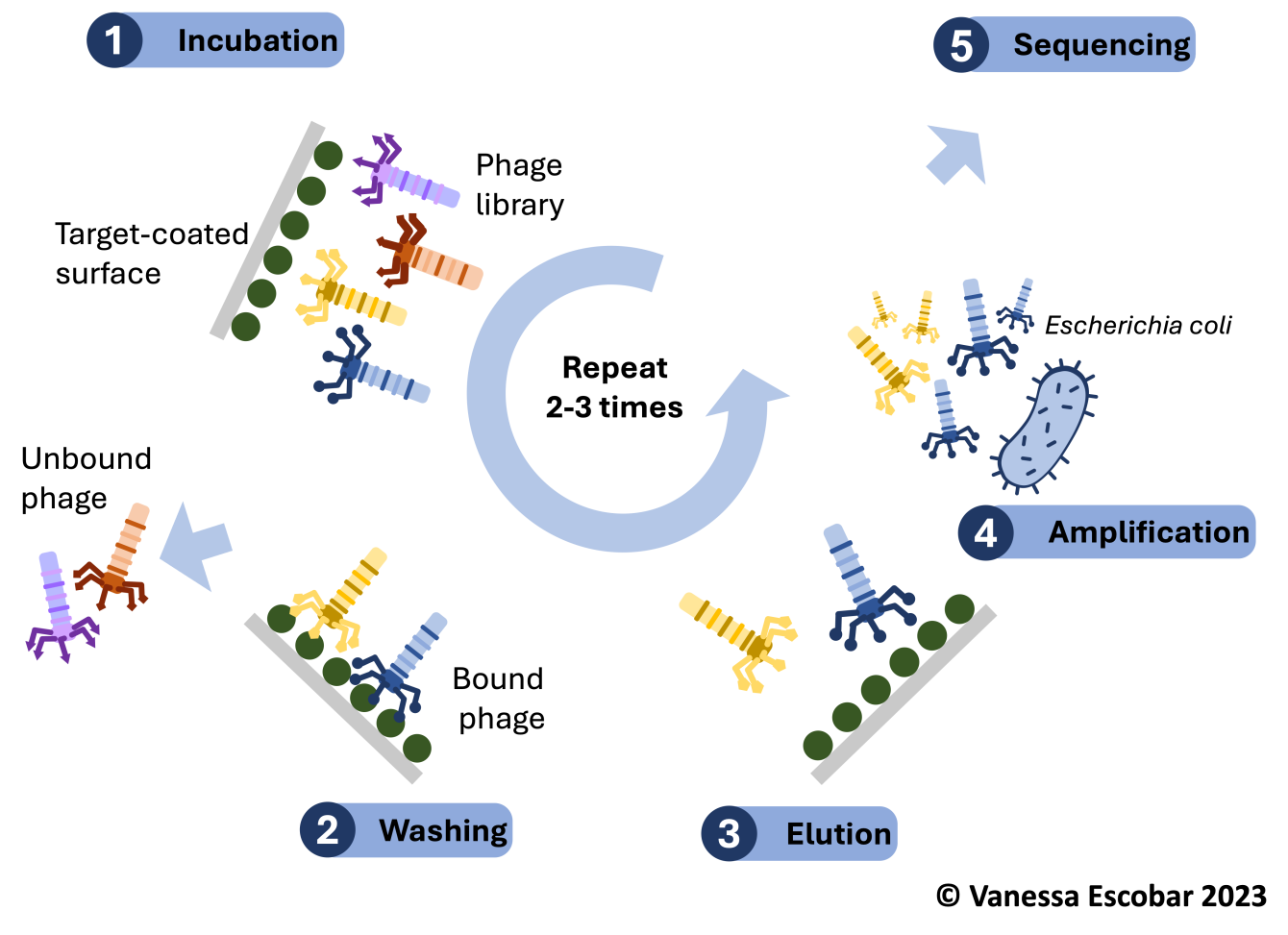
Le phage display est une technique d'évolution dirigée qui permet de sélectionner et d'optimiser des protéines (fragments d’anticorps) ou des peptides ayant des propriétés souhaitées. Dans cette méthode, des bibliothèques de phages (virus qui infectent les bactéries) sont génétiquement modifiées pour exprimer une variété de peptides ou de protéines à leur surface. En exposant ces bibliothèques à une cible spécifique, seuls les phages présentant des peptides ou des protéines ayant une forte affinité pour la cible sont retenus. Ces phages sont ensuite amplifiés et soumis à des cycles successifs de sélection, améliorant progressivement les interactions souhaitées. Le processus de mutation et de sélection reproduit les principes de l'évolution naturelle, mais de manière accélérée et contrôlée en laboratoire, permettant ainsi de créer des molécules ayant des fonctions précises pour des applications en biotechnologie et en médecine. De façon générale, tous les dispositifs de détection (bio-capteurs miniaturisés, senseurs, …) nécessitent le développement de molécules qui reconnaissent une cible particulière avec une grande affinité et spécificité.
Collaborations :
- Recherche d’anticorps vis-à-vis de topologies ADN télomériques humaines (ADN G4, i-motif)
Eric Defrancq, Thomas Lavergne, Département de Chimie Moléculaire – Université Grenoble Alpes
Jérôme Dejeu, Institut Femto-ST, département MN2S, équipe Nano2BIO – Université de Franche Comté
Dennis Gomez, Institut de pharmacologie et de biologie structurale – CNRS Toulouse - Recherche de peptides vis-à-vis de composés organiques volatils (conception nez électronique)
Yanxia Hou et Arnaud Buhot, Systèmes Moléculaires et nanoMatériaux pour l'Énergie et la Santé (SyMMES) CEA – CNRS Grenoble - Recherche de peptides pour la conception de « biosenseurs »
Yanxia Hou, SyMMES CEA – CNRS Grenoble
Jasmina Vidic, MICALIS UMR1319 INRAE – Université Paris-Saclay
Carole Chaix, Institut des Sciences Analytiques ISA – Université Claude Bernard Lyon - Recherche d’anticorps pour l’imagerie moléculaire
Jakub Toczek, Laboratoire Radiopharmaceutiques Biocliniques – Université Grenoble Alpes, Inserm U1039
Jean-Luc Lenormand, Laboratoire Translationnelle et Innovation en Médecine et Complexité (TIMC) – Université Grenoble Alpes
- Imprimer
- Partager
- Partager sur Facebook
- Partager sur X
- Partager sur LinkedIn