- Imprimer
- Partager
- Partager sur Facebook
- Partager sur X
- Partager sur LinkedIn
Recrutement
Offre de thèse financée 2025-2028
Supervision : Hidde de Jong et Noël Scaramozzino
Contexte
Le projet de doctorat sera réalisé au sein de l'équipe MICROCOSME du Centre Inria de l'Université de Grenoble Alpes et du Laboratoire Interdisciplinaire de Physique (LIPhy, CNRS/UGA) sous la supervision conjointe de Hidde de Jong et Noël Scaramozzino dans le cadre du projet MuSiHC soutenu par le PEPR B-BEST. MICROCOSME et le LIPhy offrent un environnement de recherche interdisciplinaire favorisant des interactions étroites entre mathématiciens appliqués, microbiologistes, informaticiens, ingénieurs de contrôle et biophysiciens.
Objectifs
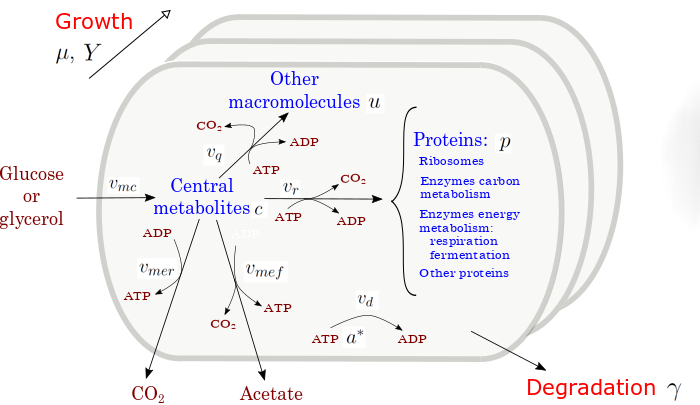
Le project MUlti-SIze Hybrid Cell Models (MuSiHC) vise à développer de nouvelles approches hybrides pour la modélisation des cellules et des bioréacteurs pour la production de composés à valeur ajoutée. En particulier, le projet développera une boîte à outils de modèles hybrides de différentes tailles, combinant une description mécaniste avec des composants AI/ML [1], afin d'obtenir des simulations de cellules et de bioréacteurs plus fiables. Comme preuve de concept, le projet se concentrera sur Escherichia coli en tant que plateforme pour la bioproduction de 1,3-propanediol (1,3-PDO), un composé de grande valeur avec de vastes applications dans l'industrie chimique.
Le projet de doctorat proposé concerne le développement de modèles dynamiques à petite échelle [2,3] pour optimiser la production de 1,3-PDO par E. coli, impliquant des tâches telles que la formulation et la réduction du modèle, l'exécution d'expériences en mini-bioréacteur pour l'étalonnage du modèle, l'utilisation des modèles pour identifier les conditions de production optimale de métabolites, et le test expérimental de ces conditions. Le projet de thèse implique une collaboration active avec d'autres partenaires de MuSiHC à l'INRAE (Jean-Loup Faulon, Wolfram Liebermeister) et à l'Institut de biotechnologie de Toulouse (César Arturo Aceves Lara). Au-delà de l'application spécifique de MuSiHC, le projet vise à identifier des principes généraux pour le développement et la validation de modèles à petite échelle de systèmes de production biotechnologique.
[1] Faure, L., Mollet, B., Liebermeister, W., & Faulon, J. L. (2023). A neural-mechanistic hybrid approach improving the predictive power of genome-scale metabolic models. Nature Communications, 14(1):4669. https://doi.org/10.1038/s41467-023-40380-0
[2] Baldazzi, V., Ropers, D., Gouzé, J. L., Gedeon, T., & de Jong, H. (2023). Resource allocation accounts for the large variability of rate-yield phenotypes across bacterial strains. eLife, 12:e79815. https://doi.org/10.7554/eLife.79815
[3] Wortel, M. T., Noor, E., Ferris, M., Bruggeman, F. J., & Liebermeister, W. (2018). Metabolic enzyme cost explains variable trade-offs between microbial growth rate and yield. PLoS Computational Biology, 14(2):e1006010. https://doi.org/10.1371/journal.pcbi.1006010
Principales activités
Le projet de doctorat est un projet interdisciplinaire impliquant à la fois le développement de modèles mathématiques décrivant le système biologique et le travail expérimental pour calibrer et valider les modèles :
- Réduction d'un modèle cinétique à moyenne échelle à un modèle à petite échelle et à cellule entière de la production de 1,3-propanediol (1,3-PDO) par Escherichia coli, en utilisant des méthodes de réduction développées précédemment et en s'inspirant de modèles existants d'allocation de ressources à petite échelle.
- Réalisation d'expériences avec des souches d'E. coli sélectionnées sur une plateforme interne de mini-bioréacteurs afin d'obtenir des données (croissance, expression génétique, concentrations de métabolites) pour l'étalonnage du modèle.
- Utilisation d'une combinaison d'approches d'optimisation et de simulation pour identifier les conditions maximisant la production de 1,3-PDO.
- Validation des conditions de fonctionnement optimales prédites en réalisant les expériences correspondantes en mini-bioréacteur, y compris la quantification de la production de 1,3-PDO.
Conditions
Les candidats intéressés doivent idéalement avoir une certaine expérience de la modélisation mathématique des systèmes biologiques et/ou des travaux de laboratoire en microbiologie, mais nous sommes ouverts aux étudiants ayant de bons résultats académiques et motivés par une recherche interdisciplinaire dans divers domaines (microbiologie, biologie mathématique, bioinformatique, écologie, biophysique, ...).
Date de début
Automne 2025
Contact and candidatures
Si vous souhaitez en savoir plus sur ce poste, veuillez contacter Hidde de Jong (hidde.de-jong inria.fr (hidde[dot]de-jong[at]inria[dot]fr)) ou Noël Scaramozzino (natale.scaramozzino
inria.fr (hidde[dot]de-jong[at]inria[dot]fr)) ou Noël Scaramozzino (natale.scaramozzino univ-grenoble-alpes.fr (natale[dot]scaramozzino[at]univ-grenoble-alpes[dot]fr)).
univ-grenoble-alpes.fr (natale[dot]scaramozzino[at]univ-grenoble-alpes[dot]fr)).
Télécharger
Contact
Hidde de Jong
Equipe BIOP
hidde.de-jong inria.fr (hidde[dot]de-jong[at]inria[dot]fr)
inria.fr (hidde[dot]de-jong[at]inria[dot]fr)
Noël Scaramozzino
Equipe BIOP
natale.scaramozzino univ-grenoble-alpes.fr (natale[dot]scaramozzino[at]univ-grenoble-alpes[dot]fr)
univ-grenoble-alpes.fr (natale[dot]scaramozzino[at]univ-grenoble-alpes[dot]fr)
- Imprimer
- Partager
- Partager sur Facebook
- Partager sur X
- Partager sur LinkedIn