- Imprimer
- Partager
- Partager sur Facebook
- Partager sur X
- Partager sur LinkedIn
Développement et analyse de modèles individus-centrés pour l’étude de la variabilité de la croissance microbienne
Aline Marguet, Eugenio Cinquemani
Non permanent·es: Claudia Fonte Sanchez (postdoctorante), Eugène Ferragu (doctorant)
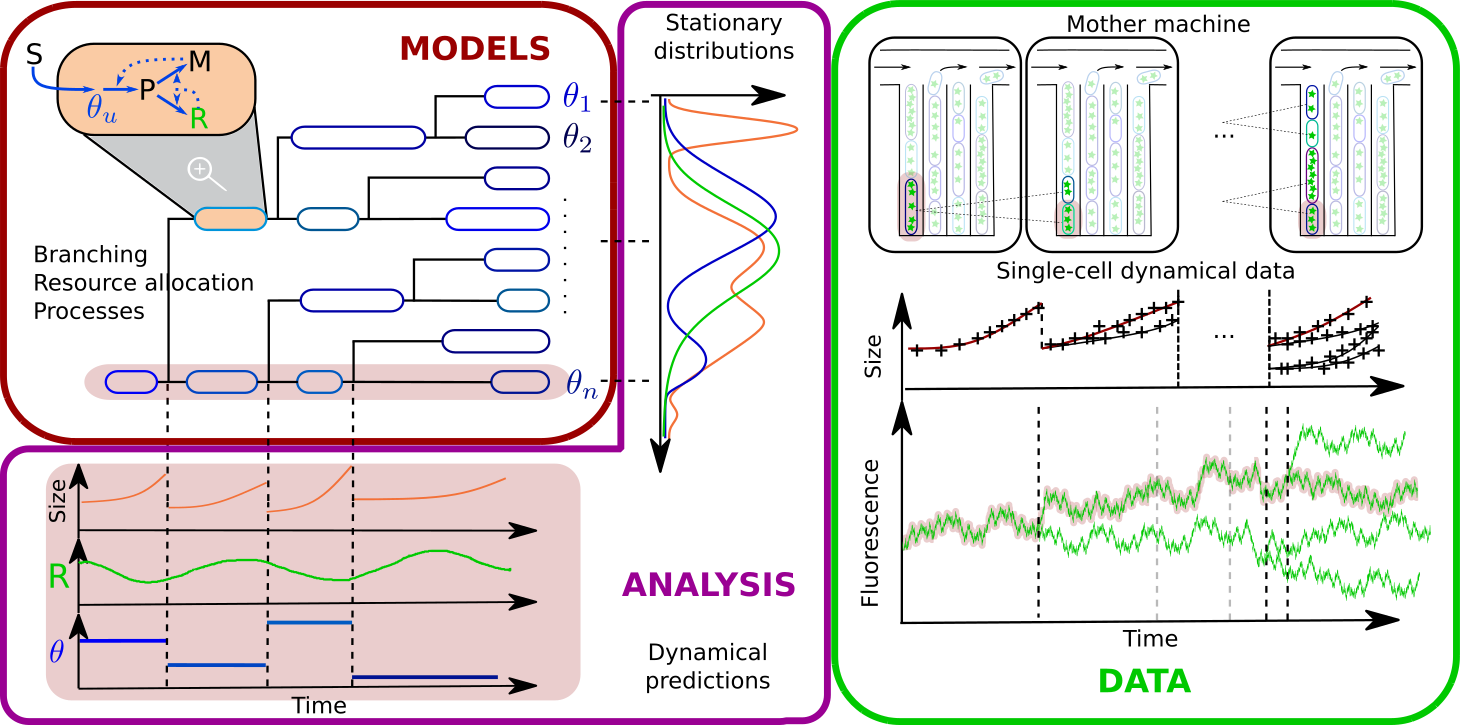
L'étude des populations de cellules à l'échelle individuelle a déjà permis la découverte de phénomènes importants tels que l'existence de phénotypes différents dans des populations isogéniques. Les nouvelles techniques expérimentales nous donnent maintenant accès à des mesures temporelles de la croissance et de l'expression génique à l'échelle individuelle, sous forme de distributions de probabilité (cytométrie de flux) ou d'arbres permettant de retracer les relations de parentés entre les cellules (usage combiné de la microscopie et des dispositifs microfluidiques). La richesse de ces données permet d'étudier quantitativement la variabilité phénotypique, à l'aide de modèles à l'échelle de la cellule individuelle. En particulier, le développement de tels modèles prenant en compte la généalogie est une étape importante pour l'étude de l'apparition de la variabilité.
À l'aide d'outils mathématiques variés, tels que les processus structurés branchants ou les modèles à effets-mixtes, nous étudions les mécanismes à l'origine de la variabilité cellulaire, tels que l'héritabilité, le partitionnement du contenu cellulaire à la division, ou la variabilité des stratégies d'allocation de ressources entre les cellules.
- Imprimer
- Partager
- Partager sur Facebook
- Partager sur X
- Partager sur LinkedIn