- Imprimer
- Partager
- Partager sur Facebook
- Partager sur Twitter
- Partager sur LinkedIn
- Share url
Allocation de ressources chez les bactéries
Johannes Geiselmann, Natale Scaramozzino, Yves Markowicz
Pour qu’une bactérie puisse s’adapter efficacement à un environnement changeant, elle doit allouer les ressources disponibles (la nourriture) de façon aussi optimale que possible. Dans l’illustration ci-contre, une source de carbone (S) est transformée en précurseurs (P, essentiellement des acides aminés) qui seront utilisés pour faire deux types de macromolécules : la machinerie de réplication d’expression génique (GEM) et les composants du métabolisme et des protéines structuraux (MM). La croissance est maximale lorsque l’allocation des ressources aux différents composants de la cellule est optimale. Nous construisons des modèles mathématiques de cette allocation de ressources (en collaboration quotidienne avec l’équipe Microcosme) et nous testons expérimentalement les prédictions des modèles (par exemple, le projet Maximic).
Les expériences mesurent le comportement de bactéries sauvages, mais nous construisons également des souches très spécifiques (biologie synthétique), comportant des modifications génomiques pour la mesure (marquages avec différentes protéines fluorescentes) ou la modification du comportement (délétion ou modification du contrôle d’expression génique). Ainsi, nous avons, par exemple, construit une souche dans laquelle l’expression de l’ARN polymérase est sous contrôle externe. Dans la même souche nous avons introduit des gènes codant pour la production d’un produit d’intérêt biotechnologique (X). Nous explorons les stratégies permettant de maximiser la production de X.
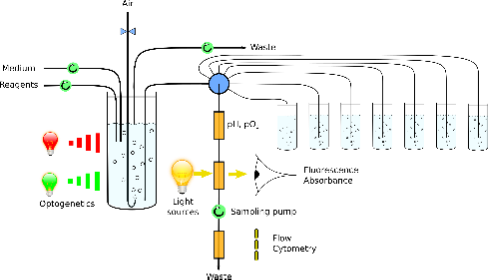
Pour mener ces expériences, nous avons construit un système de mini-bioréacteurs multiplexés permettant de contrôler les paramètres de croissance et de mesurer en temps réel des spectres d’absorbance et de fluorescence de la population ou dans les cellules uniques par cytométrie. L’expression génique peut être contrôlée par optogénétique.
Nous étudions non seulement l’optimisation de la croissance ou de la production d’une souche particulière de bactéries, mais nous élaborons également des systèmes de coopération entre différentes espèces. Les projets sont menés sur des consortia de bactéries du même type (E. coli) ou entre espèces très différentes, par exemple dans le projet CtrlAB., toujours en interaction étroite avec l’équipe Microcosme de l’INRIA.
Régulation de la production des curli et formation de biofilm
Stéphan Lacour
Nous recherchons les déterminants génétiques de l'expression et de la sécrétion des curli. Les curli sont de fins fimbriae agrégés présents à la surface des cellules des entérobactéries et responsables de la colonisation du substrat, première étape de la formation du biofilm. Nous complétons le réseau de régulation contrôlant la transcription et la traduction des opérons csg. En particulier, nous étudions l'expression du régulateur CsgD, qui est essentiel pour l'expression des gènes de structure curli (csgBA), mais aussi pour la production de cellulose et la répression des gènes flagellaires de classe 2 et 3. Plus important encore, nous voulons identifier l'interaction ARN-ARN qui contrôle la traduction de CsgD et expliquer l'expression de curli en fonction de la température.
Collaborations
Corinne Dorel, Université de LyonAkira Ishihama, Hosei University, Tokyo, Japan
- Imprimer
- Partager
- Partager sur Facebook
- Partager sur Twitter
- Partager sur LinkedIn
- Share url